Resultado Proyecto
Marcadores inmunológicos y genéticos asociados a infecciones latentes o patentes causadas por Mycobacterium avium subsp. Paratuberculosis
Referencia: RTA2014-00009-C02-02. Organismo financiador: Ministerio de Economía y Competitividad. Instituto Nacional de Investigación y Tecnología Agraria y Alimentaria (INIA). Cofinanciado por el Fondo Europeo de Desarrollo Regional (FEDER). Importe: 83.000 €. Duración: 2015-2019.
 |
 |
 |
Equipo investigador
Rosa Casais Goyos. SERIDA
José Miguel Prieto. SERIDA
Javier Amado Fernández. Laboratorio Sanidad Animal
Equipo técnico
Roxana González Álvarez. SERIDA
Resultados y conclusiones
La paratuberculosis (PTB) es una enteritis granulomatosa crónica, causada por Mycobacterium avium subsp. paratuberculosis (MAP), con graves repercusiones económicas en las explotaciones productoras ganaderas. El objetivo global de este proyecto es mejorar el control de la PTB bovina mediante la identificación de nuevos marcadores inmunológicos (biomarcadores) capaces de distinguir las distintas formas lesionales en las que se manifiesta la PTB bovina, así como, la identificación de marcadores genéticos que nos permitan seleccionar hospedadores con genotipo asociado a la susceptibilidad/resistencia a padecer un tipo específico de forma lesional.
Creación de un banco de muestras
Como punto de partida para abordar los objetivos planteados en el proyecto se creó un banco de muestras de sangre completa, suero, heces y tejido intestinal de vacas de raza frisona de 26 explotaciones localizadas en el Principado de Asturias. Las muestras de animales sacrificados proceden de una ganadería de aproximadamente 100 vacas frisonas con clínica de PTB (N=53), del rebaño experimental de leche del SERIDA (n=4) y de vacas frisonas sacrificadas en matadero (n=36). Las muestras de animales vivos (aprox. n= 300) proceden del seguimiento anual de PTB (periodo 2016-2018) realizado en la explotación de vacas frisonas (aprox. 100) con clínica de PTB y de una ganadería libre de PTB (muestras control, n=61). Las muestras se caracterizaron mediante cultivo bacteriológico y PCR a tiempo real de heces y tejidos, ELISA para la detección de anticuerpos específicos frente a MAP, y análisis histopatológico. Los animales sacrificados se clasificaron en base a la presencia/ausencia y al tipo de lesiones histológicas presentes en su tejido intestinal en cuatro grupos: focal, multifocal, difuso y sin lesiones. La clasificación histopatológica fue utilizada como técnica de referencia para estudios posteriores (transcriptómica, genómica, validación de ELISAs, etc).
Marcadores inmunológicos
Con el objeto de identificar nuevos biomarcadores de infección temprana y de progreso de la enfermedad se realizó análisis transcriptómico por RNAseq de sangre total y válvula ileocecal de vacas frisonas con lesiones focales (n=5) o difusas (n=5) y vacas control sin signos de infección detectables (n=3). El número de genes diferencialmente expresados se muestra en la Figura 1.
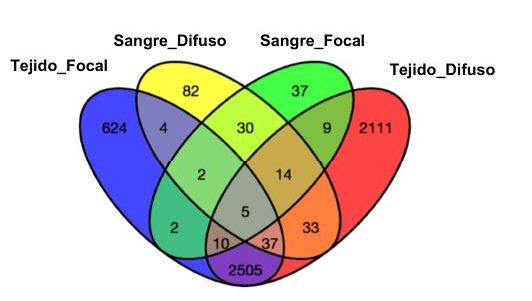
Figura 1. Diagrama de Venn mostrando los cambios de expresión génica (específicos de cada grupo lesional con respecto al control y comunes entre distintos grupos lesionales).
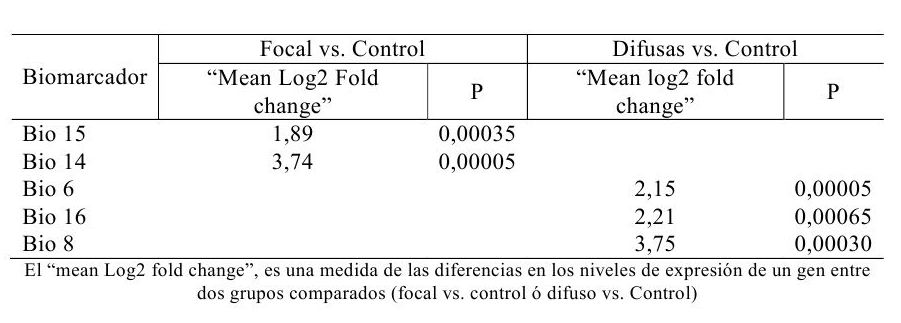
En base a los niveles de sobre-expresión (“log2 fold change”) de los genes en sangre periférica, la función de las proteínas que codifican y la disponibilidad de ELISAs comerciales específicos para su detección se seleccionaron 5 biomarcadores (Tabla 1) para su posterior validación utilizando muestras de referencia.
Tabla 1. Expresión diferencial de genes de biomarcadores bovinos seleccionados mediante análisis transcriptómico por RNA-Seq de sangre periférica de vacas de raza frisona con lesiones histológicas de tipo focal y difuso frente animales control sin lesiones.
Marcadores genéticos
Se determinó el perfil genético mediante genotipado de “single nucleotide polymorphisms” (SNPs) de todas las vacas de la ganadería asturiana con alta prevalencia de PTB (6%). Se establecieron, en base a asociaciones genéticas aditivas entre 5 de esos SNPs, grupos genéticos de bajo riesgo (LOWIN), con riesgo a desarrollar infecciones latentes (LATIN), patentes (PATIN) y animales no incluidos en los grupos anteriores (AVERIN). Se estableció una asociación entre los distintos grupos de riesgo y los ensayos diagnósticos de PTB. Las lecturas del ELISA de anticuerpos del grupo PATIN (65%) fueron significativamente más altas que las del grupo de riesgo AVERIN (15,97%), LATIN (2,11%) y LOWIN (3,27%).
Conclusiones
En este proyecto hemos identificado: 1) Marcadores genéticos para la selección de vacas resistentes a la enfermedad en programas de mejora genética y 2) Marcadores inmunológicos para el desarrollo de métodos de diagnóstico para la detección temprana de animales con infecciones subclínicas. La selección de animales resistentes y la utilización de métodos de diagnóstico más sensibles para detectar la PTB podrían contribuir de forma decisiva al control de la enfermedad en las explotaciones ganaderas.