Resultado Proyecto
Identificación de polimorfismos en el cromosoma Y de pequeños rumiantes para su utilización en filogenia
Referencia: CGL2005-03761/ BOS. Organismo financiador: Ministerio de Ciencia e Innovación. Importe: 96.985 €. Duración: 2005-2008.
Equipo investigador
Luis José Royo Martín SERIDA
Félix Mª Goyache Goñi SERIDA
Isabel Álvarez Fernández SERIDA
Lucía Pérez Pardal MICINN (Becaria)
Juan Pablo Gutiérrez García Universidad Complutense de Madrid
Equipo técnico
Iván Fernández Suárez SERIDA
Carmen Rincón Hernández SERIDA
Entidades Colaboradoras
Asociación del Corzo Español (ACE)
Asociación de Criadores d’Oveya Xalda d’Asturies (ACOXA)
Asociación de Criadores de Cabra de Raza Bermeya (ACRIBER)
Resultados
El presente proyecto tuvo como finalidad el desarrollo de una batería de marcadores específicos del cromosoma Y de pequeños rumiantes domésticos y salvajes, que puedan ser útiles en estudios filogenéticos y evolutivos. Se testaron 44 marcadores microsatélites, localizados en la región del cromosoma Y específica del macho en rumiantes domésticos, de la siguiente forma: a) se comprobó que sólo amplificaban en ADN de machos; b) se verificó su transmisión clónica de padres a hijos; c) se evaluó la facilidad y seguridad en el genotipado en diversas condiciones de PCR con el uso de diferentes polimerasas y d) se analizó su polimorfismo. En la especie bovina se han identificado cinco microsatélites polimórficos que presentaron una o dos bandas por individuo, locus y reacción de PCR, incluyéndose en dos reacciones multiplex para su genotipado. Estos marcadores demostraron tener una potencia estadística limitada para la realización de estudios filogenéticos. Los marcadores encontrados son capaces de identificar los tres haplogrupos del cromosoma Y de ganado bovino (Y1, ganado de la Europa atlántica; Y2, ganado de la Europa continental; e Y3, ganado cebú); hay una proporción importante de haplotipos que no son fácilmente asignables a la subespecie B. taurus o a la subespecie B. indicus. En todo caso, la batería de marcadores identificada permite conocer la diversidad genética existente dentro de subespecie. Como resultado adicional del proyecto, cabe resaltar el estudio de los marcadores multibanda (Interspersed multilocus microsatellites). Éstos son aquellos loci que amplifican más de una banda de diferente tamaño en el mismo individuo con una sola reacción PCR y pareja de cebadores y devienen de la naturaleza repetitiva y no recombinante del cromosoma Y. Distintas bandas pueden estar presentes o no en diferentes individuos, lo que define su polimorfismo, pudiendo tratarse estadísticamente esta información si se asume el modelo de mutación de alelos infinitos (infinite-site model). Se identificaron 12 marcadores polimórficos multibanda en ganado bovino. El análisis de repetibilidad en el genotipado permitió seleccionar dos como los más informativos. La Figura 1 resume la información proporcionada por esos marcadores que suman 48 sitios polimórficos. La imagen es compatible con un modelo de expansión de poblaciones a partir de dos procesos de domesticación (Y2 bovino e Y3 cebuino) y uno de introgresión (Y1); los tiempos de divergencia entre haplogrupos son compatibles con las estimaciones de fechas de la domesticación.
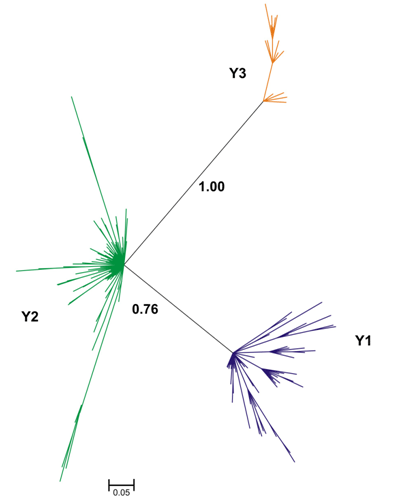
Figura 1. Análisis Bayesiano de dos marcadores multibanda sobre 453 individuos bovinos realizado mediante el programa MrBayes 3.1. El haplogrupo Y1 se muestra en azul, el haplogrupo Y2 se muestra en verde y el haplogrupo Y3 se muestra en naranja. La confianza estadística de separación del haplogrupo Y3 con los correspondientes a la subespecie B. taurus es del 100% , mientras que la separación entre los dos haplogrupos de ganado europeo es del 76%. La mayor diversidad haplotípica del haplogrupo Y1 sería compatible con una introgresión de uro europeo en el ganado doméstico.
En la especie caprina, se identificaron cuatro microsatélites polimórficos específicos de macho, dos de ellos multibanda, que sumaron 38 sitios polimórficos. De forma similar a lo encontrado en la especie bovina, la información de los marcadores multibanda seleccionados confirmó la existencia de dos haplogrupos en el cromosoma Y caprino (C1 y C2; Pidancier et al., 2006), aunque uno de ellos (C2) parece haber experimentado un intenso cuello de botella durante su formación lo que indicaría que podría ser resultado de una primera domesticación. El haplogrupo C1, presente fundamentalmente en la Península Ibérica y Oeste de África, tiene una mayor diversidad pudiendo resultar de una introgresión posterior de machos salvajes.
En ganado ovino los resultados fueron menos significativos, aunque se identificó un nuevo microsatélite multibanda polimórfico. Éste permitió identificar una familia haplotípica principal en el cromosoma Y de esta especie, que incluye el muflón (Ovis orientalis) y que resultaría ser la correspondiente al cromosoma Y ovino resultado de una primera domesticación. El resto de los haplotipos estarían más representados en ovejas asiáticas o británicas. Este hecho constituye una fuerte base para buscar SNPs filogenéticamente informativos. Esta labor se llevará a cabo en el proyecto CGL- 2008-03949.