Resultado Proyecto
Heterogeneidad ambiental y adaptabilidad en respuesta a la sequía en colecciones clonales de Pinus pinaster
Referencia: RTA2007-00084-00-00. Organismo financiador: Instituto Nacional de Investigación Agraria y Alimentaria (INIA). Importe: 129.700 €. Duración: 2007-2009.
Equipo investigador
Juan Pedro Majada Guijo. SERIDA
Isabel Feito Díaz. SERIDA
Tania Velasco Conde. INIA (Becaria)
Angelo Kidelman Dantas. SERIDA
Ricardo Alía Miranda. CIFOR/INIA
Ismael Aranda García. CIFOR/INIA
Celia Martínez Alonso. CETEMAS (Alta 2009)
Avance de resultados
Objetivo
Disponer de recursos vegetales estructurados y caracterizados en Pinus pinaster para el desarrollo de programas de conservación y mejora.
Avance de resultados
Para generar un banco clonal (CLONAPIN) que cubra la diversidad de la especie Pinus pinaster, se ha partido de material genético que forma parte de la población base del programa de mejora genética de Pinus pinaster, iniciado recientemente por el CIFOR INIA y SERIDA, así como de material procedente de poblaciones marginales con interés en conservación. A partir de estas poblaciones se ha desarrollado un programa de clonación, con una estructura genética que comprende más de 900 genotipos replicados en un banco clonal, y que representa la mayor parte de la variabilidad natural de esta especie. Previamente fue definido un procedimiento de miniesquejado para esta especie, el cual ha sido utilizado para amplificar los genotipos seleccionados hasta generar un stock clonal con al menos 10 copias por planta.
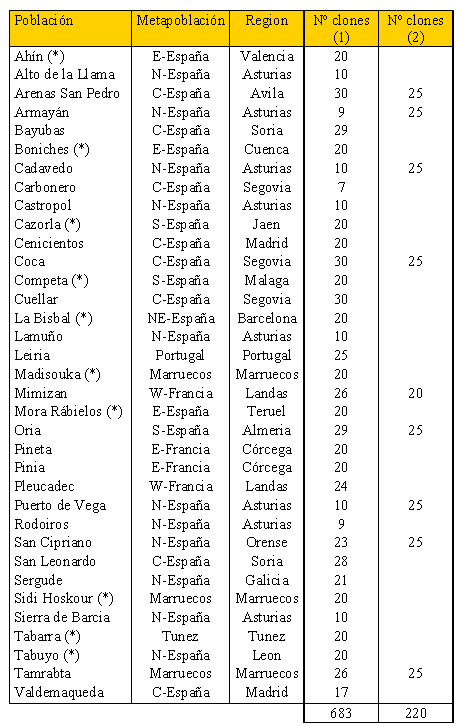
Para el establecimiento de un banco clonal de referencia se seleccionaron 35 poblaciones de Pinus pinaster (Tabla 1). La amplificación en cascada se inició con un único clon ("ortet", plántula de semilla). El banco presenta diferente estructura en sus materiales. Así, en el caso de los clones procedentes de las poblaciones indicadas con un asterisco (ver Tabla 1), no se conoce el pedigrí de sus progenitores, mientras que el resto de los clones procede siempre de familias seleccionadas fenotípicamente en campo. En este último caso, el "ortet" original se mantiene desde 2004 en campo, formando parte de la población base de mejora. Los clones (2) (Tabla 1) están constituidos por cinco "ortets" de medios hermanos de cinco familias de cada población. Esta colección de referencia se está utilizando en proyectos nacionales e internacionales.
Tabla 1. Banco clonal con las siguientes metapoblaciones: Centro-España (8), Este-España (3), Noreste-España (1), Norte-España (11), Sur-España (3), Este-Francia (2), Oeste-Francia (2), Portugal (1), Marruecos (3) y Tunez (1). (1) Clones con un genotipo por familia y presentes en la población base en campo exceptuando (*). (2) Clones con cinco genotipos clonados por familia.