Resultado Proyecto
Caracterización del gen CXCR4 bovino y su promotor: filogenia en la tribu bovini, detección de selección y asociación con la tripanotolerancia
Referencia: AGL2011-27585. Organismo financiador: Ministerio de Ciencia e Innovación. Importe: 121.000 €. Duración: 2011-2014.
 |
Equipo investigador
Félix Mª Goyache Goñi. SERIDA
Isabel Álvarez Fernández. SERIDA
Albano Beja-Pereira. CIBIO Universidade do Porto
Shanyuan Chen. CIBIO Universidade do Porto
Amadou Traoré INERA
Hamidou H. Tamboura INERA
Equipo técnico
Iván Fernández Suárez. SERIDA
Entidad Colaboradora
Conference des Responsables de Recherche Agronomique Africains / West and Central African Council for Agricultural Research and Development (CORAF/WECARD).
Resultados y conclusiones
Se han analizado un total de 8994 pares de bases (pb) correspondientes a zonas exónicas (codificantes de proteínas) e intrónicas (no codificantes) de los genes CXCR4, TICAM1 e INHBA, de los que existe evidencia científica previa de su relación con la tripanotolerancia bovina, en 96 muestras bovinas, incluyendo bovino europeo, africano tripanotolerante, africano tripanosusceptible y cebú africano y asiático, así como otros miembros de la Tribu bovini: Bos frontalis (gaur), Bos grunniens (yak), Bison bonasus (bisonte europeo), Bison bison (bisonte americano), Syncerus caffer (búfalo rojo), Connochaetes taurinus (ñu), Ovis aries (oveja), y h) Capra hircus (cabra). Asimismo, se han analizado los 99 pb del exón 9 del gene bovino ARHGAP15 por su posible importancia en la determinación del carácter.
Se diseñaron oligonucleótidos, utilizando la secuencia del genoma bovino UMD3.1 como referencia, para la secuenciación de fragmentos nucleotídicos de tamaño variable entre los 400 y 500 pb mediante la tecnología 454 FLX Titanium Sequencer (Roche, Branford, EE.UU.) en la empresa Lifesequencing S.L. (Valencia). La tecnología 454FLX exige oligonucleótidos de tipo “fusion” en la forma: Primer 1: (5′) Adaptor A + 10-mer Multiplex IDentifier (MID) + Key + [Forward primer] (3′); Primer 2: (5′) Adaptor B + Key + [Reverse primer] (3′) (siendo Adaptor las siguientes secuencias: A: 5′-CCATCTCATCCCTGCGTGTCTCCGAC-3′; y B: 5′-CCTATCCCCTGTGTGCCTTGGCAGTC-3′, and Key: 5′-TCAG-3′). Esta tecnología permite multiplexar diferentes muestras en una sola carrera de secuenciación. Las secuencias brutas obtenidas (archivos sff) se han remitido al repositorio publico NCBI Short Read Archive (BioProject nº SRP044810). Los archivos sff se han analizado mediante los paquetes bioinformáticos de acceso público sff2fastq, CutAdapt, Tophat2 y Samtools para obtener un archivo vcf que incluía los sitios polimórficos respecto de las secuencias base del genoma bovino UMD3.1.
El polimorfismo identificado ha resultado ser muy bajo: en el gen CXCR4 solo se han validado seis sitios polimórficos de los que cuatro eran intrónicos y los restantes se localizaban en el exón 2, uno de ellos (posición BTA2:61584846) que producía una sustitución aminoacídica (198V→A) en el dominio transmembrana de la proteína; tanto en el gen INHBA como en el gen TICAM1 se identificaron ocho mutaciones que producían cambios aminoacídicos, aunque solo dos de esas mutaciones (correspondientes al gen TICAM1) se identificaron en más de un cromosoma; en el exón 9 del gen ARHGAP15 bovino se encontraron 3 mutaciones previamente descritas y una nueva en ganado cebuino.
Todas las mutaciones que se consideraron posibles candidatas para la tripanotolerancia bovina se encontraron en todos los grupos de ganado bovino analizados, desde el ganado europeo al cebú asiático. Ninguna de ellas fue privativa del ganado bovino autóctono del oeste de África, lo que excluye su consideración como causa subyacente de la tripanotolerancia bovina. En consonancia con la baja variabilidad genética encontrada, análisis filogenéticos (bayesianos y de grafos) no permitieron encontrar una estructura genética en las secuencias analizadas que permitiera sugerir un papel decisivo de alguna de las zonas genómicas analizadas en la determinación del carácter. Aunque una de las causas que podrían explicar la situación encontrada podría ser la selección purificadora, los test de neutralidad calculados excluyeron esa hipótesis. Sólo en el caso del gen CXCR4 se encontró un bloque de secuencia en desequilibrio de ligamiento (Figura 1). Un análisis pormenorizado permitió conocer que este desequilibrio se debía fundamentalmente a las poblaciones cebuinas del oeste de África, que se encuentran en continuo flujo genético con las poblaciones de ganado bovino autóctono de esa área continental.
Las conclusiones que pueden obtenerse de los análisis realizados son las siguientes:
a) Ninguno de los genes analizados, sobre los que existía una importante evidencia científica sobre su posible papel en la determinación genética de la tripanotolernacia bovina presentó mutaciones privativas del ganado bovino autóctono del oeste de África, lo que cuestiona su identificación como genes candidatos para el carácter o, al menos, su actuación como genes mayores sobre el mismo.
b) El escenario genético descrito es compatible con una determinación genética aditiva de baja heredabilidad para el carácter. La gran identidad genética encontrada en los genes y grupos bovinos analizados explicaría esta situación.
c) En la medida en que la existencia de desequilibrio de ligamiento esencial para la utilización de técnicas de mapeo genético y estudios de asociación, nuestros resultados sugieren que futuros estudios de asociación para la tripanotolerancia en poblaciones no experimentales deberían realizarse no utilizando exclusivamente individuos de razas bovinas autóctonas del oeste de África sino, fundamentalmente, individuos de las poblaciones cebuinas del oeste de África.
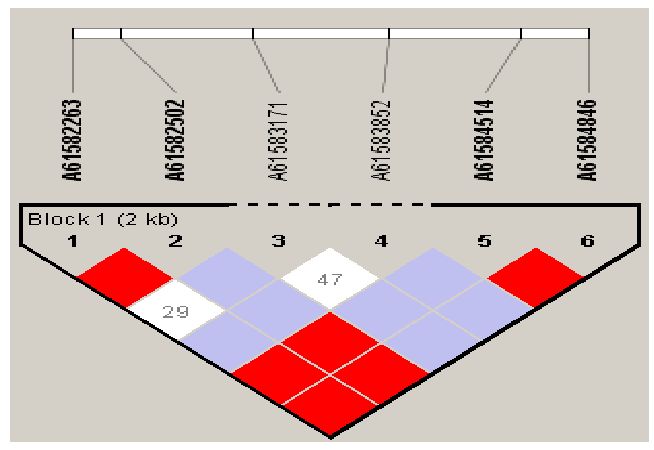
Figura 1. Mapa de desequilibrio de ligamiento (r2) calculado sobre seis mutaciones validadas identificadas en el gen CXCR4 bovino. Los números en negrita corresponden a la posiciones genómicas que se encuentran en desequilibrio.