Resultado Proyecto
Identificación, caracterización genética y conservación de levaduras no-Saccharomyces aisladas en sidras asturianas. Evaluación de sus actividades enzimáticas
Referencia: RM2006-00008-00-00. Organismo financiador: Instituto Nacional de Investigación y Tecnología Agraria y Alimentaria. Importe: 25.680 €. Duración: 2006-2009.
Equipo investigador
Belén Suárez Valles SERIDA
Rosa María Pando Bedriñana SERIDA
Equipo técnico
Ana Lastra Queipo SERIDA
Resultados y conclusiones
Se abordó la caracterización genética, tanto a nivel de especie como de cepa, de las levaduras no-Saccharomyces, aisladas de mostos y sidras asturianas. Asimismo, se realizó la caracterización fenotípica de todas las cepas mediante la evaluación de sus actividades enzimáticas.
En primer lugar, se llevaron a cabo las identificaciones a nivel de especie de las levaduras (438 cepas) mediante el análisis de la región ribosómica 5,8S y los espaciadores intergénicos ITS1 e ITS2. La posterior digestión de los amplificados con las enzimas de restricción Cfo I, Hae III, Hinf I permitió la clasificación de las cepas en: Hanseniaspora valbyensis (46%), Hanseniaspora osmophila (10%), Metschnikowia pulcherrima (13%), Pichia guilliermondii (2 cepas), Candida parapsilopsis (10%) y Dekkera bruxellensis (4%). Para el resto de las levaduras fue necesario la utilización de un cuarto enzima (Dde I) cuyos fragmentos de restricción agruparon a las 77 cepas restantes como pertenecientes a la especie Hanseniaspora uvarum.
A continuación, se realizó la evaluación cualitativa de las actividades enzimáticas en las cepas genéticamente identificadas) sobre medios sólidos (Figura 1).
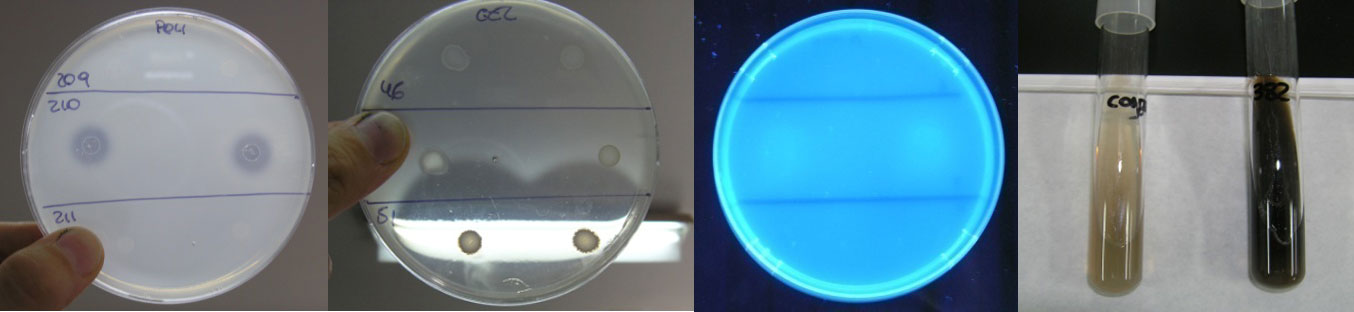
Figura 1. Visualización de las actividades enzimáticas, de izquierda a derecha: poligalacturonasa, proteasa, β-xilosidasa y β-glucosidasa.
La producción de las enzimas pectolíticas fue observada en menor extensión: la actividad pectín-liasa no fue encontrada en ninguno de los aislados y la actividad poligalacturonasa tan sólo se halló en ocho cepas pertencientes al género Hanseniaspora y en una levadura identificada como D. bruxellensis. La actividad β-glucosidasa está presente en cepas de todas las especies, destacando el elevado número de cepas de las especies D. bruxellensis (83%), M. pulcherrima (73%) y H. uvarum (49%) con dicha actividad. La actividad β-xilosidasa tan sólo fue detectada en: 11 cepas del género Hanseniaspora, cinco aislados clasificados como M. pulcherrima y en una de las cepas P. guilliermondii. La hidrólisis de la gelatina la produjo el 14% de las cepas, destacando esta capacidad en el 49% de las levaduras M. pulcherrima; contrariamente, esta actividad no está presente en P. guilliermondii y en D. bruxellensis. Dos cepas pertenecientes a las especies H. uvarum y M. pulcherrima presentaron la capacidad proteolítica además de las dos glícosidásicas (Tabla 1).
Tabla 1. Levaduras con varias actividades enzimaticas
| Actividades enzimáticas | H. valbyensis | H. uvarum | M. pulcherrima | H. osmophila | C. parapsilosis | P. guilliermondii | D. bruxellensis |
| Poligalacturonasa y β-glucosidasa | 1 | 1 | |||||
| β-glucosidasa y proteasa | 5 | 12 | 21 | 6 | 2 | ||
| β-glucosidasa y β-xilosidasa | 1 | ||||||
| β-glucosidasa, β-xilosidasa y proteasa | 1 | 1 |
Para la detección de la variabilidad genética, a nivel de cepa, se utilizó el método RAPD-PCR con el oligonucleótido OPA-03 y los microsatélites ((GAC)5, (GTG)5, RM13). Las dos cepas pertenecientes a la especie P. guilliermondii se caracterizaron mediante RFLP del ADN mitocondrial, obteniendo perfiles de restricción distintos para ambas cepas.
Con los distintos cebadores se obtuvieron diferentes patrones de amplificación, mostrando sus perfiles genéticos una gran diversidad tanto en el número de polimorfismos como en sus tamaños moleculares. En todas las especies estudiadas se observó que el microsatélite (GAC)5 generó un mayor número de bandas, con la excepción de las cepas identificadas como D. bruxellensis, para las que el oligonucleótido OPA-03 produjo mayor polimorfismo. Se obtuvo una gran variabilidad, entre los perfiles genéticos, en función de la especie y del cebador utilizado (Tabla 2).
Tabla 2. Variabilidad genética de las distintas especies.
| M. pulcherrima | C. parapsilopsis | H. osmophila | H. uvarum | H. valbyensis | D. bruxellensis | |
| Cebador |
Variabilidad (%)
|
|||||
| OPA03 | 58,2 | 52,4 | 48,8 | 80,3 | 38,0 | 77,8 |
| RM13 | 96,4 | 54,8 | 78,0 | 89,4 | 36,4 | 44,4 |
| GTG | 72,7 | 78,6 | 65,9 | 87,9 | 80,2 | 94,4 |
| GAC | 90,9 | 88,1 | 87,8 | 72,7 | 92,0 | 83,3 |
| OPA-03 y RM13 | 98,2 | 85,7 | 92,7 | 98,5 | 73,8 | 88,9 |
| TODOS | 98,2 | 97,6 | 95,1 | 98,5 | 98,4 | 88,9 |
Para encontrar las posibles similitudes entre cepas se utilizó el paquete estadístico NTSY, aplicando el algoritmo de Jaccard y visualizando su agrupamiento mediante análisis cluster. No se encontraron agrupamientos comunes entre cepas con los distintos oligonucleótidos ensayados.
Las similitudes obtenidas con el OPA-03, entre las 18 cepas de la especie Dekkera, agrupan en tres clusters a siete cepas con una similitud del 100%, mientras que el microsatélite (GAC)5 aglutina a cinco cepas en dos grupos con el mismo nivel de agrupamiento (Figura 2).
Dekkera bruxellensis
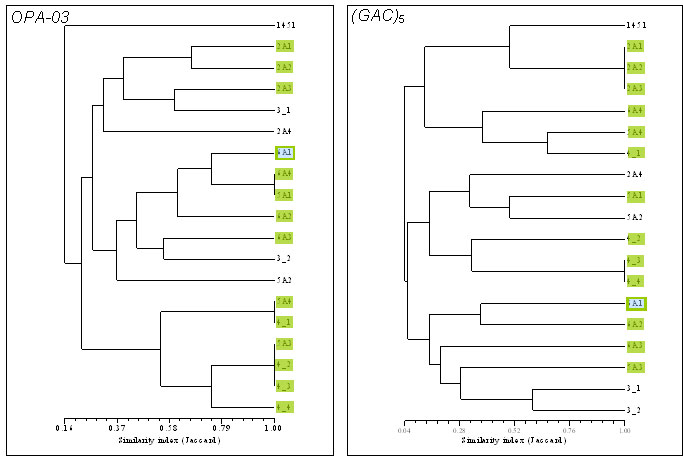
Figura 2. Agrupamientos de similitud para las cepas D. bruxellensis (ref: 1451, cepa testigo de la Colección Española de Cultivos Tipo). Las cepas con fondo verde mostraron actividad β-glucosidasa, cepas con fondo azul y borde verde poseen, además, actividad poligalacturonasa.
Los cebadores OPA-03 y RM13 generaron mayores agrupamientos entre las cepas identificadas como C. parapsilopsis. Para ambos oligonucleótidos la variabilidad detectada fue próxima al 50%. El dendograma obtenido con el cebador RM13 da lugar a cinco grupos de levaduras que poseen una similitud del 100%. No obstante, las cepas que presentan iguales perfiles genotípicos muestran disimilitud respecto a las actividades enzimáticas. Para los cebadores (GAC)5 y (GTG)5 se obtuvieron menores agrupamientos y, por lo tanto, una mayor variabilidad genética.
El cebador que generó un mayor agrupamiento entre las cepas M. pulcherrima fue el OPA-03, sin embargo, las cepas que se agruparon con una similitud del 100% desarrollaron distintas actividades enzimáticas. El cluster que presentó una mejor correlación con la caracterización fenotípica fue el obtenido con la combinación de los cebadores OPA-03 y RM13.
En el caso de las levaduras del género Hanseniaspora se obtuvieron resultados similares para las tres especies estudiadas. Cuando se analizaron los grupos naturales generados entre las cepas, para cada cebador individualmente, de nuevo, dieron lugar a disimilitudes con sus actividades enzimáticas. Como era de esperar, las representaciones cluster obtenidas con la combinación de los cuatro cebadores originaron una mayor diversidad y permitió una mejor correlación entre las actividades enzimáticas y los perfiles de caracterización genética de las cepas.
La colección de levaduras no-Saccharomyces se mantiene duplicada (-80º C y -196º C), preservando la diversidad genética de las levaduras autóctonas.