Resultado Proyecto
Uso combinado de marcadores polimórficos de evolución rápida y lenta en la filogenia del cromosoma Y de pequeños rumiantes domésticos
Referencia: CGL2008-03949/BOS. Organismo financiador: Instituto Nacional de Investigación y Tecnología Agraria y Alimentaria. Importe: 130.438 €. Duración: 2008-2011.
Equipo investigador
Luis José Royo Martín. SERIDA
Félix Mª Goyache Goñi. SERIDA
Isabel Álvarez Fernández. SERIDA
Lucía Pérez Pardal. MICINN (becaria)
F. Abel Ponce de León. Universidad de Minnesota
Equipo técnico
Iván Fernández Suárez. SERIDA
Carmen Rincón Hernández. SERIDA
Entidades Colaboradoras
Asociación de Criadores d’Oveya Xalda d’Asturies (ACOXA)
Asociación de Criadores de Cabra de Raza Bermeya (ACRIBER)
Justificación
El análisis de la diversidad del cromosoma Y ha demostrado su utilidad para el establecimiento de las relaciones filogenéticas y evolutivas en la especie humana. Sin embargo, en poblaciones animales, y especialmente en pequeños rumiantes, apenas existen estudios filogenéticos basados en la diversidad genética del cromosoma Y.
Objetivo
El presente proyecto pretende resolver el mayor problema de los microsatélites específicos del cromosoma Y, esto es, su gran tasa de mutación (aproximadamente 1/500 años); ello da lugar a homoplasias que dificultan el establecimiento de relaciones filogenéticas claras y el cálculo de tiempos de divergencia dentro y entre especies.
Se persigue detectar mutaciones puntuales (SNPs) en fragmentos de ADN de la zona específica masculina de pequeños rumiantes (MSY) que puedan tener un alto significado evolutivo utilizable en filogenia, tanto en secuencias codificantes como no codificantes.
Este objetivo general se complementará con los siguientes objetivos concretos: a) identificación de SNPs en secuencias codificantes de fragmentos localizados en la zona MSY de ovinos, caprinos y bovinos (como control positivo); b) identificación de SNPs en secuencias codificantes de genes localizados en la zona MSY a partir de las secuencias homólogas conocidas fundamentalmente en la especie humana); c) desarrollo de protocolos diagnósticos basados en RT-PCR acoplada a sondas fluorescentes para el genotipado de los SNPs identificados en las diferentes especies de pequeños y grandes rumiantes; d) establecimiento de relaciones filogenéticas intra e inter específicas en las especies de rumiantes estudiadas para la caracterización del flujo genético paterno; y e) estimar los tiempos de divergencia de las líneas paternas identificadas en el cromosoma Y de las diferentes especies y entre los cromosomas Y de las diferentes especies.
Resultados y conclusiones
Se presentan los resultados del genotipado con los marcadores identificados en el cromosoma Y de las especies de pequeños rumiantes domésticos:
Ovino: Se han identificado dos SNPs y dos microsatélites en la región no recombinante del cromosoma Y ovino. Se han diseñado protocolos específicos para genotipar 548 machos de 68 poblaciones de ovejas domésticas y muflones de África, Asia, América y Europa (incluyendo la raza autóctona asturiana Xalda).
Caprino: Se han identificado nueve SNPs y tres microsatélites localizados en la región no recombinante del cromosoma Y caprino. Se ha diseñado protocolos específicos pare genotipar 300 machos de 30 poblaciones de cabras de África, Asia y Europa (incluyendo la raza autóctona asturiana Bermeya).
Ovino
El genotipado de 548 machos de poblaciones de ovejas domésticas y muflones originó tres haplogrupos y 28 haplotipos. Los haplogrupos son combinaciones diferentes de SNPs, y los haplotipos combinaciones de alelos de los microsatélites dentro de cada haplogrupo. En este caso los haplogrupos comparten haplotipos, lo que nos indica que o bien la división que observamos en haplotipos no tiene estructura filogenética, o que la información que proporcionan los marcadores no es suficiente para inferir la filogeografía de los ovinos domésticos.
La figura 2 recoge los 28 haplotipos diferentes encontrados en un árbol filogenético tipo "Network" (Figura 1).
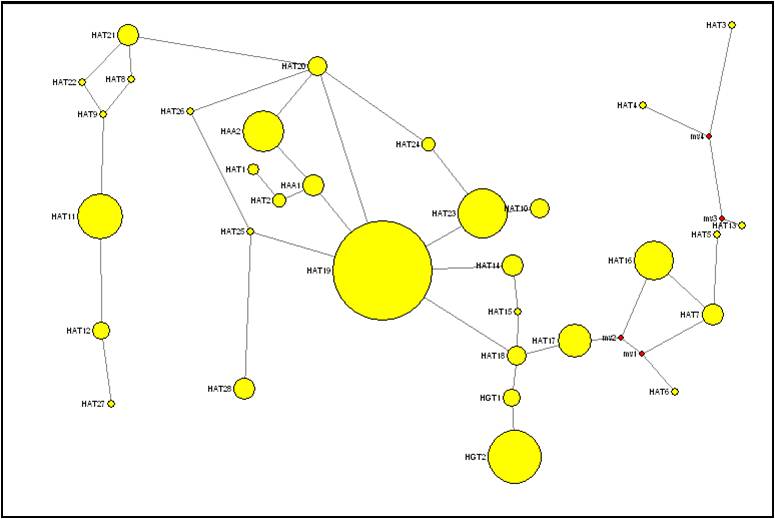
Figura 1: Imagen "Network" de los 28 haplotipos identificados en el cromosoma Y de los ovinos.
Caprino
Con los genotipos obtenidos las muestras se agrupan en siete haplogrupos diferentes (Figura 2) y 17 haplotipos. Los haplogrupos no comparten haplotipos, lo que indica cierta consistencia en la división y separación de los haplogrupos, y por tanto una estructura filogenética (Figura 2).
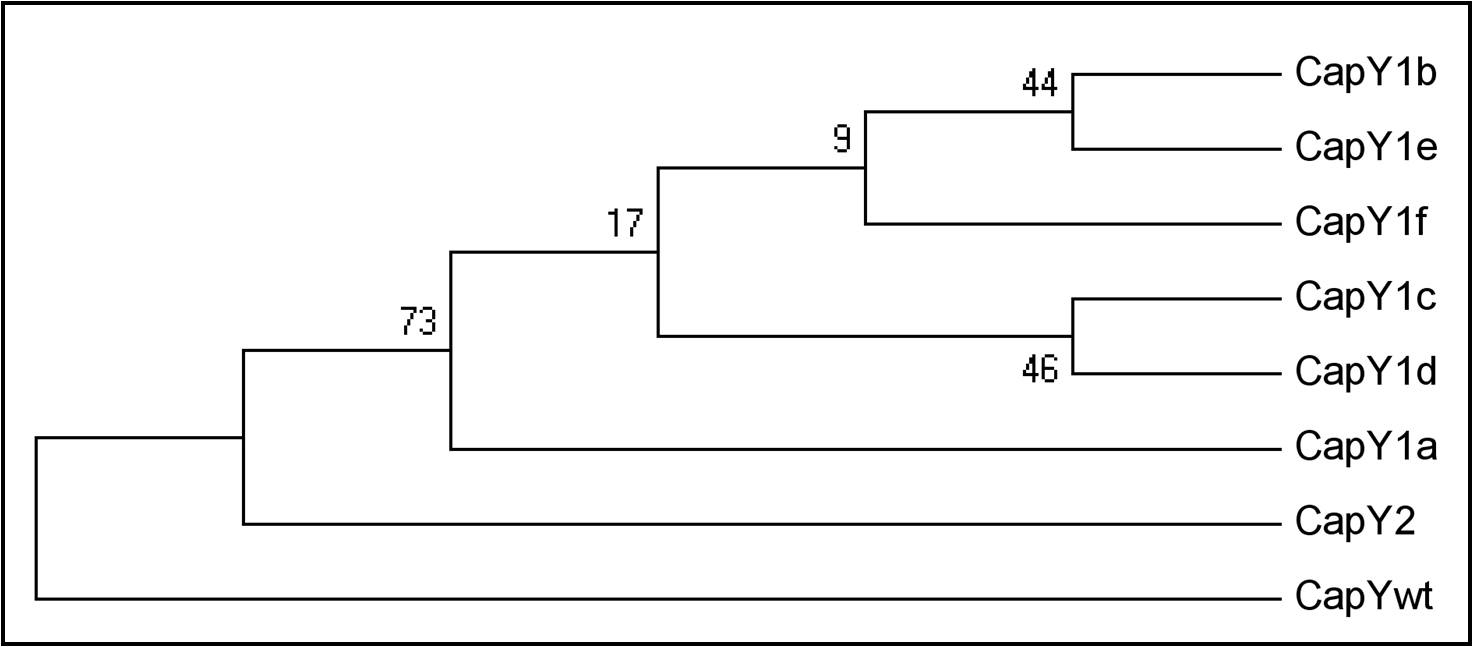
Figura 2: Árbol filogenético UPGMA de los haplogrupos identificados en el cromosoma Y caprino. Se ha usado un macho de cabra montés de Gredos como referencia (CapYwt).
Los genotipos de los 300 machos caprinos domésticos (más uno silvestre usado como referencia), se utilizaron para construir un árbol filogenético "Network". En la Figura 3 se muestran los 17 haplotipos identificados en el cromosoma Y.
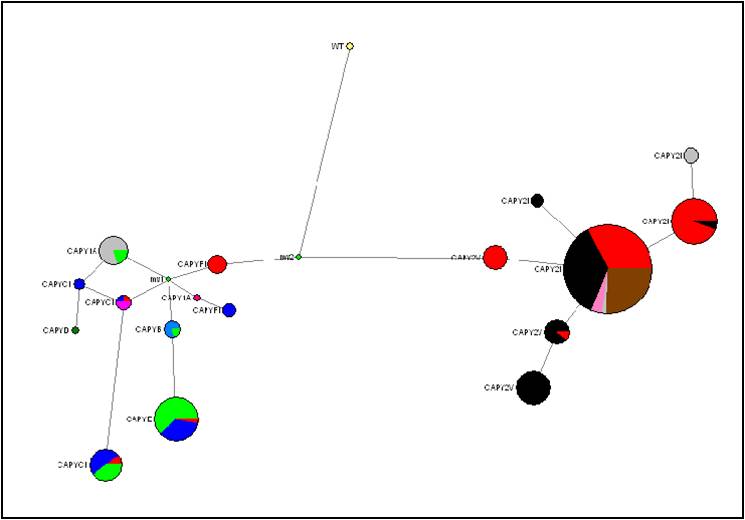
Figura 3: Imagen "Network" de los haplotipos del cromosoma Y caprino.
Se observa cierta estructuración geográfica, siendo mayoritario el haplogrupo CapY2 (derecha) en las poblaciones africanas, mediterráneas y península arábiga (negro, gris y marrón), y los haplogrupos Y1 (izquierda) en poblaciones europeas (azul y verde). La península ibérica (rojo) parece tener una posición intermedia entre ambas, apareciendo tanto haplotipos CapY2 como CapY1.