Resultado Proyecto
Obtención de información de coascendencia molecular para optimización de la conservación de la variabilidad genética
Referencia: RZ2004-00007-C02-02. Organismo financiador: Instituto Nacional de Investigación y Tecnología Agraria y Alimentaria. Importe: 52.200 €. Duración: 2004-2008.
Equipo investigador
Félix Mª Goyache Goñi SERIDA
Luis José Royo Martín SERIDA
Isabel Álvarez Fernández SERIDA
Lucía Pérez Pardal MICINN (Becaria)
Juan Pablo Gutiérrez García Universidad Complutense de Madrid
Equipo técnico
Iván Fernández Suárez SERIDA
Carmen Rincón Hernández SERIDA
Entidades Colaboradoras
Asociación de Criadores d’Oveya Xalda d’Asturies (ACOXA)
Asociación de Criadores de Ganado Ovino de Raza Colmenareña
Asociación de Criadores de Ganado Ovino de Raza Rubia de El Molar
Associació de Ramaders de l’Ovella de Raça Mallorquina
Avance de resultados
El proyecto tiene como objetivo principal la implantación de una metodología de monitorización de los libros genealógicos de cuatro razas ovinas españolas en riesgo para seleccionar aquellos reproductores cuya utilización asegure la representación genética de los animales fundadores en la siguiente generación. Los distintos libros genealógicos presentan grandes diferencias en contenido informativo. Por ello, se utiliza la raza Xalda como unidad sobre la que testar las metodologías propuestas para su posterior aplicación en las otras razas. Sobre esta raza se ha testado un escenario que incluía el objetivo de incrementar la frecuencia del genotipo ARR/ARR, eliminar el alelo VRQ y reducir la frecuencia del alelo ARQ (a propuesta de ACOXA), que parece estar asociado a mayor riesgo de padecimiento de Encefalitis Espongiforme Bovina en la oveja. Se identificaron los animales nacidos durante el último año y seleccionados para recría y se calcularon las pérdidas de variabilidad genética, respecto de los animales adultos de cada sexo, cuando se incluía o no el genotipo de "scrapie" como criterio de selección.
Los resultados se resumen en la Figura 1. La variabilidad genética se pierde fundamentalmente por la deriva genética causada por la selección de cada nuevo grupo de reproductores. La utilización de los genotipos de "scrapie" como criterio de selección no afecta a las hembras pero si a los machos, debido al escaso número de corderos de genotipo ARR/ARR seleccionables.
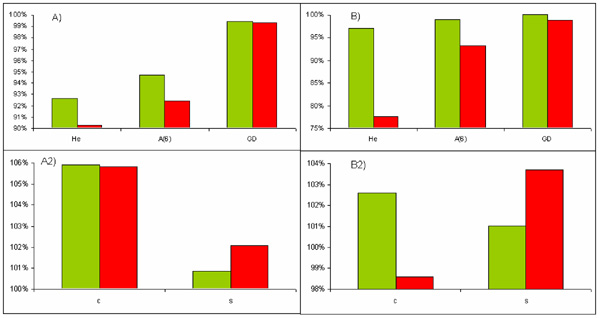
Figura 1. Porcentaje de variabilidad genética retenida considerando (barras rojas) o no el genotipo de "scrapie" como criterio de selección. La imagen A) muestra los ratios de heterocigosis esperada (He), número medio de alelos por locus (A) y diversidad genética genealógica (GD) y la A.2) la autocoascendencia esperada molecular (s) y genealógica (c), correspondientes a las hembras. Las imágenes B) y B2) proporcionan la misma información respecto de los machos.