Resultado Proyecto
Caracterización genética, evaluación y conservación de bacterias lácticas aisladas de sidras asturianas
Referencia: RM2009-00005-00-00. Organismo financiador: Instituto Nacional de Investigación y Tecnología Agraria y Alimentaria (INIA). Importe: 47.898 €. Duración: 2009-2013.
 |  |
 |
Equipo investigador
Belén Suárez Valles. SERIDA
Rosa María Pando Bedriñana. SERIDA
Equipo técnico
Mª Teresa Valderas Herrero. SERIDA
Resultados y conclusiones
En este Proyecto se abordó la identificación y caracterización por técnicas moleculares de 399 bacterias lácticas (BAL) provenientes, tanto de aislamientos realizados en distintas fases de la fermentación, en llagares asturianos, como de sidras con y sin defectos sensoriales. La caracterización tecnológica de las cepas respecto al rendimiento de la transformación maloláctica, la producción de polisacaridos exocelulares y la capacidad para generar aminas biógenas y precursores del carbamato de etilo, proporciona información útil para avanzar en el conocimiento de las bacterias lácticas.
Estudio de la diversidad genética en bacterias lácticas autóctonas mediante la aplicación de técnicas de biología molecular.
Se recuperaron y purificaron las BAL de la Colección de Cultivos Autóctonos del SERIDA, que se mantenían conservadas en medio glicerado (-80ºC), en un MRS suplementado con un 2% de concentrado de manzana (pH=5.4), incubación a 29ºC y en anaerobiosis. Todos los aislados fueron clasificados como Gram (+) y catalasa (–), observando dos tipos de morfología celular: ovoide (45%) y bacilar (55%). A las cepas con morfología ovoide se les determinó su carácter homo y heterofermentativo, el 30% mostraron un metabolismo homofementativo.
Se identificaron todas las BAL mediante secuenciación de la región del rDNA 16S, utilizando los oligonucleótidos PA y UP1-R. Las secuencias nucleotídicas de estos fragmentos fueron comparadas con las depositadas en la base de datos GenBank (NCBI). Todos los aislados se clasificaron con porcentajes de similitud >98% en nueve especies (Tabla 1). Destacar que la especie más abundante aislada durante la fermentación maloláctica fue O. oeni mientras que en sidras alteradas lo fue Lactobacillus collinoides/paracollinoides.
Tabla 1. Identificación de bacterias lácticas mediante secuenciación del ADNr 16S.
Seguidamente, se abordó la caracterización molecular de las cepas por especies, mediante el análisis de los polimorfismos obtenidos por RAPD. Se utilizó el cebador Coc para los aislados de la especie L. collinoides/paracollinoides y el oligonucleótido M13 para el resto de las especies. La aplicación del análisis de similitud entre los perfiles de amplificación, utilizando el coeficiente de similitud de Pearson’s y el método UPGMA, agrupó a los 127 aislados de la especie O.oeni en 27 clusters diferentes. Por esta técnica los aislados de origen sidrero de L. hilgardii y L. plantarum/pentosus fueron clasificados en dos perfiles de amplificación diferentes. Todos los L. diolivorans presentaron un único perfil, con la excepción de un aislado procedente de una sidra filada. En el resto de especies el número de polimorfismos fue más elevado.
Los perfiles de similitud, con el cebador Coc, para las cepas L. collinoides/paracollinoides, permitió clasificar en seis grupos los 190 aislados de origen sidrero. Uno de los clusters agrupa a 167 aislados junto con la cepa de referencia L. collinoides CECT 922, indicando que el 88% de los aislados pertenecen a la especie L. collinoides.
Evaluación del potencial biotecnológico de las bacterias láctica y su aptitud para la formación de sustancias nocivas para la salud.
Se realizó el “screening” de las BAL productoras de exopolisacáridos (EPS) y de fenotipo “ropy” en medios suplementados con 20 g/L de cada uno de los azúcares mayoritarios del mosto de manzana (Figura 1). Las especies que se revelaron como mayores productoras de EPS fueron P. ethanolidurans y P. parvulus, mientras que, las especies O. oeni y L. planatarum/pentosus fueron las menos productoras. Respecto al fenotipo “ropy” la especie P. ethanolidurans, de nuevo, fue la especie que presentó mayoritariamente este carácter. Por su origen, las BAL productoras de EPS y fenotipo “ropy” provienen de sidras filadas (Figura 2).
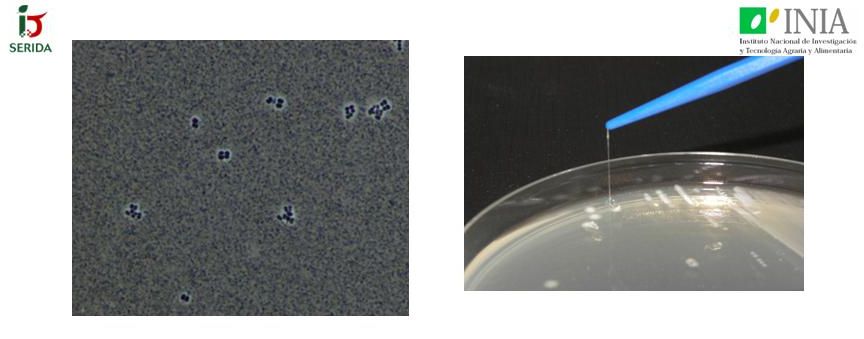
Figura 1. Producción de exopolisacáridos y fenotipo “ropy” en un aislado de L. ethanolidurans (ref:210).
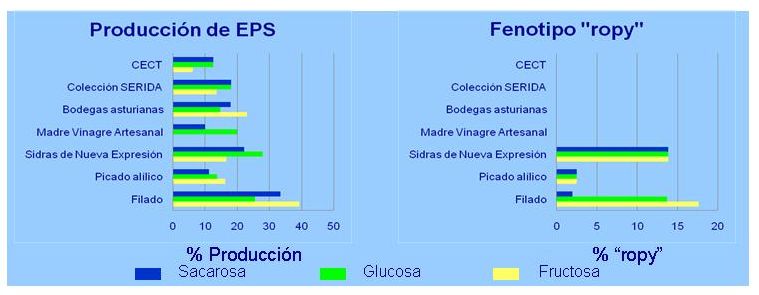
Figura 2. Porcentajes de aislados productores de exopolisacáridos (EPS) y fenotipo “ropy” en presencia de los azúcares del mosto de manzana.
En las cepas de las especies O. oeni y L. plantarum/pentosus se evaluó la aptitud para metabolizar el málico transformándolo en ácido láctico y en otros productos secundarios de la transformación maloláctica. A bajas temperaturas (15ºC), los aislados de O.oeni mostraron una mayor capacidad para transformar el ácido málico en comparación con las cepas de la especie L. plantarum.
La aptitud de las BAL para producir las aminas biógenas más significativas (histamina, tiramina, feniletilamina, cadaverina y putrescina) se evaluó, de forma cualitativa en todos los aislados, en medios sólidos.
Ningún aislado, de origen sidrero, presentó capacidad para descarboxilar la tirosina, mientras que, la arginina fue el aminoácido que presentó mayores porcentajes de descarboxilación. En estos medios ninguno de los aislados de L. plantarum/pentosus, L. rhamnosus, L. mesenteroides y P. ethanolidurans mostró capacidad para producir aminas biógenas. Todos los L. hilgardii descarboxilaron fenilalanina, ornitina, arginina y lisina. La producción de putrescina, a partir de ornitina, sólo fue detectada en dos cepas de P. parvulus y en un único aislado de las especies O. oeni y L. diolivorans (Tabla 2). En el caso de la especie L. collinoides/paracollinoides el 12% de los aislados no produjeron aminas, en torno al 40% tuvieron actividades histidina, fenilalanina y ornitina descarboxilasa, mientras que las actividades arginina y lisina descarboxilasa fueron desarrolladas por más del 60% de los aislados.
Tabla 2. Actividades descarboxilasas y formación de putrescina
| Medios sólidos |
HPLC | |||||
| Feniletilalanina | Histidina | Arginina | Lisina | Ornitina | Putrescina (g/L) |
|
| P. parvulus | ||||||
| 42 | - | - | - | - | + | 2,2 |
| 43 | - | - | - | - | + | 2,1 |
| O.oeni |
||||||
| 355 | - | - | - | - | + | 1,4 |
| L. diolivorans |
||||||
| 275 | - | - | - | - | + | 2,2 |
Paralelamente se optimizó un método de HPLC para la determinación cuantitativa de las aminas biógenas y de los aminoácidos precursores del carbamato de etilo (citrulina y ornitina). La separación cromatográfica se realizó a 43ºC, en fase reversa XTerra (150x4.6 mm, 5µm), previa derivatización automatizada de la muestra con OPA/3-MPA y detección dual: detector de fluorescencia (λ excitación= 340nm y λ emisión= 425nm) y fotodiodos (λ=338 nm). Las fases móviles fueron metanol y una reguladora de acetato sódico 40 mM y se trabajó en modo gradiente.
Se determinó la producción de aminas biógenas, en medios líquidos, de un número significativo de aislados no productores de aminas, en medio sólido, y pertenecientes a distintas especies y de aquellos aislados que mostraron actividad ornitina descarboxilasa. Los resultados cuantitativos confirmaron los obtenidos de forma cualitativa. Las cepas de L. hilgardii que mostraron actividad arginina deiminasa (ruta ADI), produjeron cantidades variables de citrulina y ornitina (Figura 3).
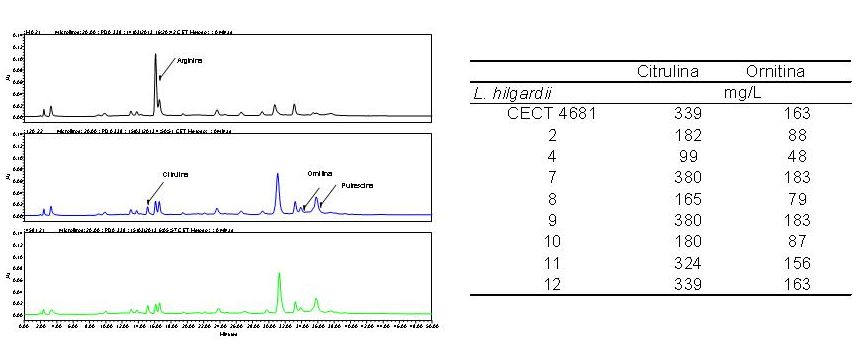
Figura 3. Producción de precursores del carbamato de etilo en cepas de la especie L. hilgardii.