Resultado Proyecto
Desarrollo de un método de diagnóstico de sarna sarcóptica basado en el uso de antígenos recombinantes de Sarcoptes scabiei y anticuerpos monoclonales. Identificación de antígenos de S. scabiei con potencial vacunal
Referencia: CIT-060000-2009-34. Organismo financiador: Ministerio de Ciencia e Innovación. Importe: 156.463 €. Duración: 2009-2011.
Equipo investigador
Rosa Casais Goyos SERIDA
José Miguel Prieto Martín SERIDA
Antonio José Sanz Fernández INGENASA
Carmén Vela Olmo INGENASA
Equipo técnico
Paloma Solano Sobrado SERIDA
Ana Camuñas Talavera INGENASA
Avance de resultados
Los resultados obtenidos son los siguientes:
1. Se ha desarrollado un ELISA de competición para el diagnóstico “multiespecie” de la sarna sarcóptica.
2. Se ha puesto a punto un modelo animal para realizar infestaciones experimentales de conejos New Zealand White con ácaros de S. scabiei procedentes de conejos silvestres infestados de forma natural (figura 1).
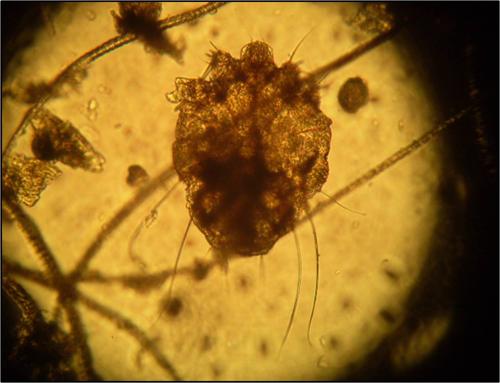
Figura 1. S. scabiei aislado a partir de conejos salvajes afectados de sarna sarcóptica.
Este modelo animal se ha utilizado para ensayar el potencial vacunal del antígeno recombinante GST-Ssλ20∆B3. En la figura 2 se muestran las jaulas utilizadas para realizar estos experimentos.

Figura 2. Bloque de jaulas utilizado para realizar las infestaciones experimentales y los ensayos vacunales.
Se comprobó que los animales vacunados con la proteína de fusión desarrollaban niveles altos de IgGs específicas mientras que los conejos inmunizados con PBS no desarrollaron anticuerpos durante la fase de vacunación (figura 3).
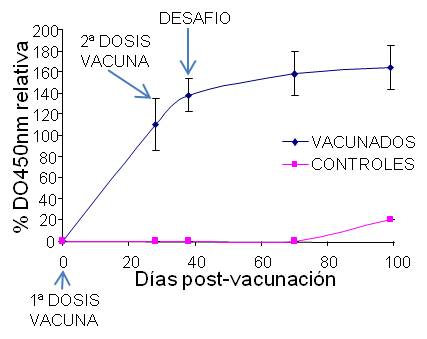
Figura 3. Evolución de la respuesta inmune a lo largo del tiempo.
Para valorar si la respuesta inmune observada protegía a los conejos frente a una infestación con S. scabiei, todos los conejos fueron desafiados con Sarcoptes. Se hizo un seguimiento semanal de la respuesta inmune, una monitorización clínica de la enfermedad y un estudio parasitológico para confirmar el origen de las lesiones observadas. Así, se comprobó que, tras el desafío, los niveles de anticuerpos de los animales vacunados siguieron aumentando para finalmente mantenerse constantes. En cuanto a la patología de la enfermedad, los animales vacunados, aunque desarrollaron lesiones típicas de sarna con cierto retraso con respecto al grupo control, la severidad de dichas lesiones alcanzó niveles similares en ambos grupos a las nueve semanas post-infección. Por lo tanto, podemos concluir que la proteína recombinante GST- sλ20∆B3 no protege a los conejos frente a la infestación con S. scabiei.
3. Dado que el antígeno GST-Ssλ20∆B3 no es un buen candidato vacunal, se procedió a la identificación de otros antígenos de Sarcoptes que, utilizados de forma individual o combinados (cocktails de antígenos) pudieran resultar más eficaces como vacunas.
Se han identificado nueve clones inmunoreactivos que se clasificaron, teniendo en cuenta su secuencia nucleotídica, en cuatro grupos (ver tabla 1). El grupo I, formado por 5 clones (Ssλ51, 52, 53, 57, 58) con secuencias derivadas de un único gen, tiene un interés especial, ya que presenta una identidad elevada con el gen de Ochletalus triseriatus OT-148 Q que codifica una proteína abundante en la saliva. Las proteínas presentes en la saliva son fundamentales para la supervivencia del ácaro, ya que contienen enzimas que intervienen en la digestión. En consecuencia, una vacuna dirigida a estas proteínas sería una diana efectiva para neutralizar este ectoparásito.
Tabla 1. Clasificación y características preliminares de los clones inmunoreactivos seleccionados.
| Grupo |
Lambda |
Clon |
Tamaño inserto (Kb) |
|
I |
ʎ51 |
5 |
1,2 |
|
ʎ52 |
9 |
1,3 |
|
|
ʎ53 |
13 |
1,25 |
|
|
ʎ57 |
24 |
1,2 |
|
|
ʎ58 |
28 |
1,3 |
|
|
II |
ʎ56 |
21 |
0,9 |
|
III |
ʎ55 |
17 |
1,1 |
|
IV |
ʎ55-A |
19 |
1,9 |
|
|
ʎ50 |
1 |
1,5 |
Se pretende continuar con esta línea de investigación mediante la producción de estos antígenos en Escherichia coli, ensayando su potencial vacunal en el modelo animal conejo/S. scabiei.

